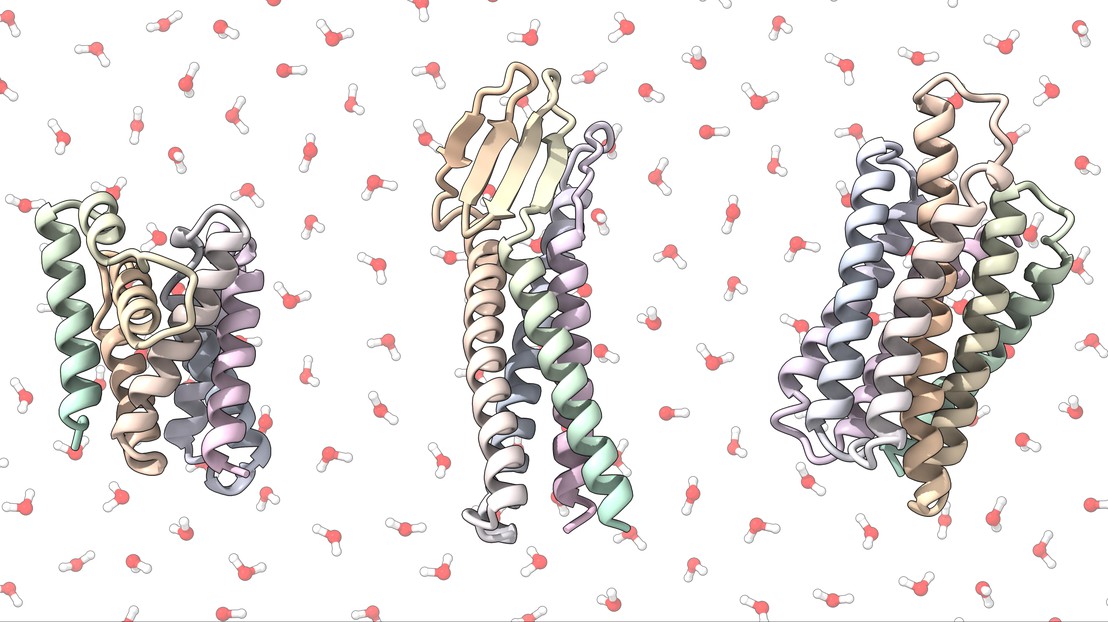
بسیاری از مسیرهای کشف دارو و آنتیبادی بر روی پروتئینهای غشای سلولی تا شده متمرکز میشوند: وقتی مولکولهای یک داروی کاندید به این پروتئینها متصل میشوند، مانند کلیدی که به داخل قفل میرود، آبشارهای شیمیایی ایجاد میکنند که رفتار سلولی را تغییر میدهند. اما از آنجایی که این پروتئین ها در لایه بیرونی سلول های حاوی لیپید تعبیه شده اند، دسترسی به آنها دشوار است و در محلول های آب گریز نامحلول هستند و مطالعه آنها را دشوار می کند.
کاسپر گوورد، دانشجوی دکترا در آزمایشگاه طراحی پروتئین توضیح میدهد: «ما میخواستیم این پروتئینها را از غشای سلولی خارج کنیم، بنابراین آنها را بهعنوان آنالوگهای بیشپایدار و محلول که شبیه پروتئینهای غشایی به نظر میرسند، اما کار با آنها بسیار آسانتر است، دوباره طراحی کردیم». و مهندسی ایمنی (LPDI) در دانشکده مهندسی.
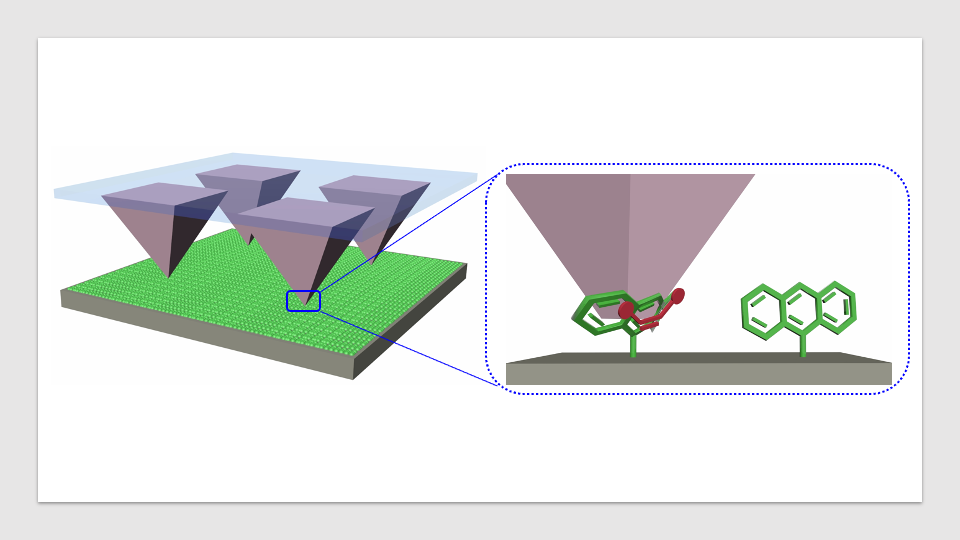
به طور خلاصه، Goverde و یک تیم تحقیقاتی در LPDI، به رهبری برونو کوریا، از یادگیری عمیق برای طراحی نسخههای محلول مصنوعی پروتئینهای غشای سلولی که معمولاً در تحقیقات دارویی استفاده میشوند، استفاده کردند. در حالی که روشهای غربالگری سنتی بر مشاهده غیرمستقیم واکنشهای سلولی به داروها و آنتیبادیهای نامزد، یا استخراج با زحمت مقادیر کمی از پروتئینهای غشایی از سلولهای پستانداران متکی است، رویکرد محاسباتی محققان به آنها اجازه میدهد تا سلولها را از معادله حذف کنند. پس از طراحی یک آنالوگ پروتئین محلول با استفاده از خط لوله یادگیری عمیق، آنها می توانند از باکتری ها برای تولید پروتئین اصلاح شده به صورت عمده استفاده کنند. سپس این پروتئین ها می توانند مستقیماً در محلول با کاندیدهای مولکولی مورد علاقه متصل شوند.
نیکلاس گلدباخ، دانشجوی دکترا، میافزاید: «ما تخمین میزنیم که تولید دستهای از آنالوگهای پروتئین محلول با استفاده از E. coli حدود 10 برابر کمتر از استفاده از سلولهای پستانداران است.
در سالهای اخیر، دانشمندان با موفقیت از شبکههای هوش مصنوعی استفاده کردهاند که از یادگیری عمیق برای طراحی ساختارهای پروتئینی جدید استفاده میکنند، برای مثال با پیشبینی آنها بر اساس توالی ورودی از بلوکهای سازنده اسید آمینه. اما برای این مطالعه، محققان به چینهای پروتئینی که از قبل در طبیعت وجود دارند علاقه داشتند. آنچه آنها نیاز داشتند نسخه قابل دسترس تر و قابل حل این پروتئین ها بود.
گوورد در ادامه افزود:ما این ایده را داشتیم که این خط لوله یادگیری عمیق را که ساختار پروتئین را پیشبینی میکند معکوس کنیم اگر ساختاری را وارد کنیم، آیا میتواند دنباله اسید آمینه مربوطه را به ما بگوید؟
برای دستیابی به این هدف، تیم از پلتفرم پیشبینی ساختار AlphaFold2 از Google DeepMind استفاده کرد تا توالیهای اسید آمینه را برای نسخههای محلول چندین پروتئین غشای سلولی بر اساس ساختار سهبعدی آنها تولید کند. سپس، آنها از دومین شبکه یادگیری عمیق، ProteinMPNN، برای بهینه سازی آن توالی برای پروتئین های کاربردی و محلول استفاده کردند. محققان از کشف این که رویکرد آنها موفقیت و دقت قابل توجهی را در تولید پروتئینهای محلول نشان داد که بخشهایی از عملکرد اصلی خود را حفظ میکرد، خوشحال بودند، حتی زمانی که برای چینهای بسیار پیچیده که تاکنون از سایر روشهای طراحی دور ماندهاند، استفاده شود.
موفقیت خاص این مطالعه، موفقیت خط لوله در طراحی یک آنالوگ محلول با شکل پروتئین شناخته شده به عنوان گیرنده جفت شده با پروتئین G (GPCR) بود که حدود 40 درصد از پروتئین های غشای سلولی انسان را نشان می دهد و یک هدف اصلی دارویی است.
ما برای اولین بار نشان دادیم که میتوانیم شکل GPCR را بهعنوان یک آنالوگ محلول پایدار دوباره طراحی کنیم. این یک مشکل طولانی مدت در بیوشیمی بوده است، زیرا اگر بتوانید آن را محلول کنید، میتوانید داروهای جدید را بسیار سریعتر و آسانتر بررسی کنید. مارتین پاچسا، دانشمند LPDI می گوید.
محققان همچنین این نتایج را اثباتی برای کاربرد خط لوله آنها در تحقیقات واکسن و حتی درمان سرطان می دانند. به عنوان مثال، آنها یک آنالوگ محلول از نوع پروتئینی به نام کلودین طراحی کردند که در مقاوم کردن تومورها به سیستم ایمنی و شیمی درمانی نقش دارد. در آزمایشهای خود، آنالوگ کلودین محلول تیم، خواص بیولوژیکی خود را حفظ کرد و وعده خط لوله را برای تولید اهداف جالب برای توسعه دارویی تقویت کرد.
Materials provided by Ecole Polytechnique Fédérale de Lausanne. Original written by Celia Luterbacher. The original text of this story is licensed under Creative Commons CC BY-SA 4.0. Note: Content may be edited for style and length.